Один из способов, с помощью которого клетки могут контролировать активность своих генов, заключается в добавлении к ДНК небольших химических модификаций, которые определяют, какие гены включены или выключены. Метильные группы являются одной из таких химических модификаций или меток. Исследователи обнаружили, что у бактерий метилирование ДНК играет роль в регуляции вирулентности, размножения и экспрессии генов. У других организмов, включая человека, метилирование ДНК необходимо для регуляции экспрессии тканеспецифических генов, которая определяет природу клетки, например, будет ли она клеткой кожи или клеткой мозга.
«Изучение метилирования ДНК является частью области эпигенетики. Оно важно, потому что помогает нам понять, почему один конкретный тип бактерий вызывает более тяжелое заболевание, чем другой, или как нормальная клетка может измениться и вызвать такие заболевания, как рак», — сказал автор-корреспондент доктор Тао Ву, доцент кафедры молекулярной генетики и генетики человека в Медицинском колледже Бейлора. Wu Lab — лаборатория эпигенетики рака. Его долгосрочная цель — преодолеть терапевтическую резистентность рака за счет лучшего понимания роли эпигенетики в этом заболевании.
У бактерий есть три различных формы метилирования ДНК. Наиболее распространенным является тот, который помечает основание ДНК или строительный блок аденина (N6-метиладенин или 6mA). Два других метят основание ДНК цитозин (N4-метилцитозин или 4mC и 5-метилцитозин или 5mC). Хотя существует множество методов изучения метилирования ДНК, некоторые из них могут эффективно картировать три типа одновременно, объяснил Ву.
«Считалось, что другие организмы, кроме бактерий, в том числе млекопитающие, в основном использовали только метилцитозиновые метки — 5mC — для регуляции активности генов. Но в 2016 году, когда я работал в Йельском университете, мы сообщили в Nature об открытии того, что ДНК 6mA также присутствует у млекопитающих», — сказал Ву. «Это открытие открыло целый ряд новых возможностей в изучении эпигенетики рака».
Традиционные методы изучения 5mC не фиксируют метилирование аденина в тканях млекопитающих. «Это побудило нас разработать новый метод для профилирования не только 6 мА, но также 4 мКл и 5 мКл», — сказал Ву.
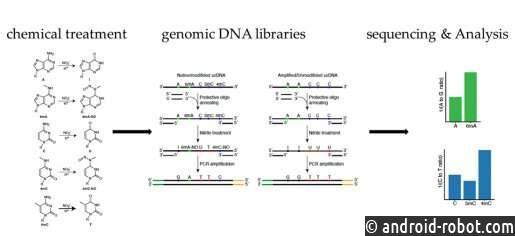
В текущем исследовании, опубликованном в журнале Genome Biology, Ву и его коллеги сообщают о разработке химического метода секвенирования для одновременного количественного определения различных эпигенетических маркеров. Их метод, называемый NT-seq, сокращенно от обработки нитритом с последующим секвенированием следующего поколения , представляет собой метод секвенирования для обнаружения нескольких типов метилирования ДНК по всему геному. Этот метод также может амплифицировать ограниченные клинические образцы, чего не могут сделать другие методы.
«Мы показываем, что NT-seq может обнаруживать 6 мА, 4 мКл и 5 мКл как в бактериальных, так и в небактериальных клетках, включая клетки млекопитающих », — сказал Ву. «По сравнению с другими методами NT-seq эффективнее, экономичнее, быстрее и имеет высокое разрешение. Некоторые из его ограничений связаны с конкретным составом некоторых геномов. В статье есть предложения о том, как компенсировать это ограничение. »
«Мы в восторге от NT-seq, — сказал Ву. «Он может раскрывать новые паттерны или мотивы метилирования ДНК, подтверждать результаты, полученные с помощью других методов, генерировать наборы данных для разработки инструментов машинного обучения для анализа метилирования и прокладывать путь к дальнейшему эпигенетическому изучению геномной ДНК 6mA в небактериальных организмах, включая исследования.